← Back
RBE | 快速算法赋能超大生物体系分子动力学模拟,助力高通量药物筛选和精准设计
2022年1月
【背景介绍】
分子动力学模拟(Molecular dynamics simulation,简称MD simulation),作为一种基于牛顿力学积分的、描述微观尺度下分子层面粒子间相互作用及动力学的计算模拟方法,在物理、化学、材料和生物等领域广泛应用,并取得巨大成就。然而目前传统的分子动力学模拟往往被限制在100万原子以下的小体系及小于10微秒的时间尺度,远不足以刻画许多真实的系统。
库仑相互作用(分子间的长程静电相互作用力计算复杂度高「O(N^2)」)是最主要的计算瓶颈,它的长程性导致计算核心之间密集通讯,大大降低使用计算机集群时的并行效率。因此,发展一种具有高精度、高速率和高可扩展性的分子模拟算法至关重要。2019年,上海交通大学自然科学学院院长金石教授与李磊教授、杜克大学刘建国教授提出的随机分批法(Random Batch Methods-RBM)成功将计算复杂度降到了N的量级,并给出了严格的误差收敛性估计。
【研究成果】
近期,上海交通大学数学科学学院徐振礼教授和洪亮课题组合作,将RBM算法赋能计算化学,构造了Random Batch Ewald(RBE)算法来计算分子间的长程静电力。该成果入选第二届“上海交通大学十大科技进展”。目前,RBE方法已申请专利(受理号202110095324.8)。
“上海交通大学十大科技进展” 是2020年上海交通大学为贯彻国家创新驱动发展战略,进一步提升科研团队产出重大科研成果的积极性,以科技创新推进学校高水平发展而设立的。
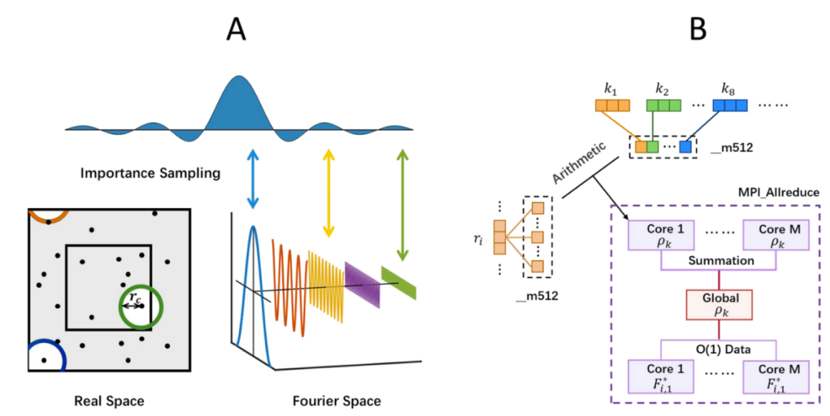
RBE算法的示意图(A)和高性能实现流程图(B)
目前该算法已应用于多个主流计算模拟软件LAMMPS、Gromacs等进行全原子分子动力学模拟。测试结果证明RBE算法在保持主流算法的精度下,能够将万核并行效率提高5倍,计算速度提高10倍。
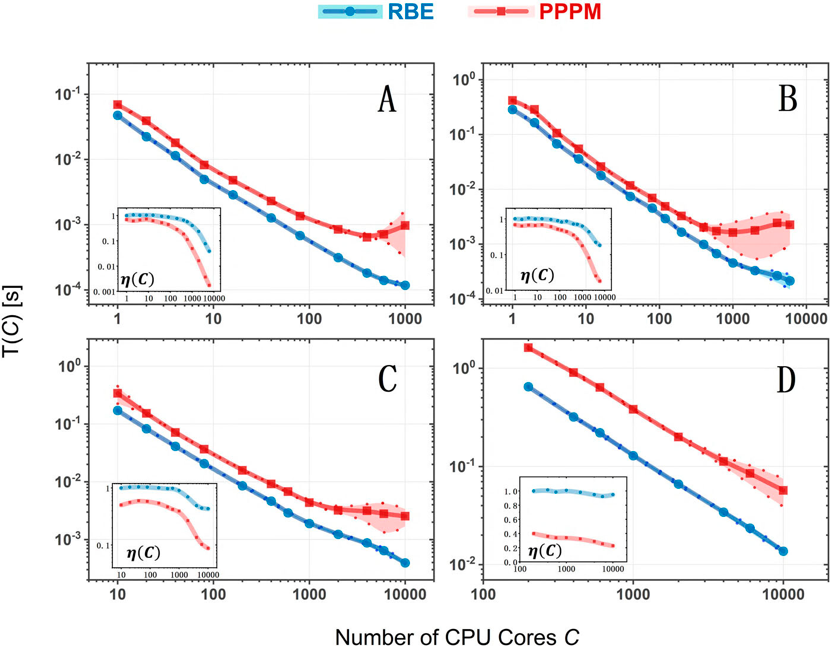
RBE算法相较PPPM算法具有更强劲的性能和超级可扩展性
此外,洪亮课题组联合上海交通大学上海应用数学中心团队在“思源一号”上完成了LAMMPS-RBE(LAMMPS-RBE是由上海交通大学上海应用数学中心团队基于LAMMPS二次开发的、完整应用RBE的自研软件)的测试。
点击此处试用:
https://docs.hpc.sjtu.edu.cn/app/engineeringscience/lammps-rbe.html?highlight=rbe
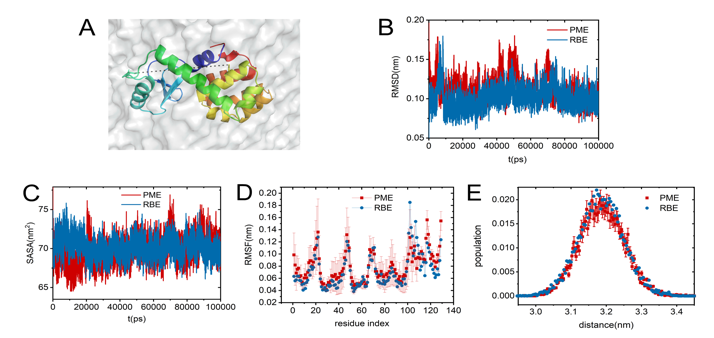
蛋白质溶液下RBE算法精确重现主流算法获得的结构、动力学及热力学信息
“思源一号”是上海交通大学全新上线的全国顶尖高性能计算机群,总计算力6千万亿次/秒,在中国高性能计算TOP 100榜单上排名12位,在中国高校高性能计算排名第一,其拥有的万核CPU对验证LAMMPS-RBE的大规模并行效率验证提供了很好的硬件条件,以下是部分测试数据。
【测试体系与结果一】
3亿个原子的全原子水体系,使用CPU核数量从1024到51200核,比较基于不同静电求解器(RBE和PPPM)的CPU时间和相对并行效率。并行效率是基于RBE方法在1024核时候的平均每核计算时间,并行效率计算方式为平均每核计算时间的相对比例。
PPPM在CPU万核开始,由于通讯开销大幅上升,计算时间反而随核数增加而下降。LAMMPS-RBE比PPPM在使用51200核时有接近三个数量级的相对并行效率提升,万核以内模拟的强扩展性在95%左右。
【测试体系与结果二】
全原子水体系, 使用CPU核数量从1到21384核,固定每个CPU核平均有4500个原子以测试基于不同静电求解器(RBE和PPPM)的CPU时间和弱可扩展性。
随着CPU核数的增加PPPM算法扩展性明显下降,1000核时下降到~10%。相比较,LAMMPS-RBE软件依旧在万核模拟中实现了接近80%的弱可扩展性。
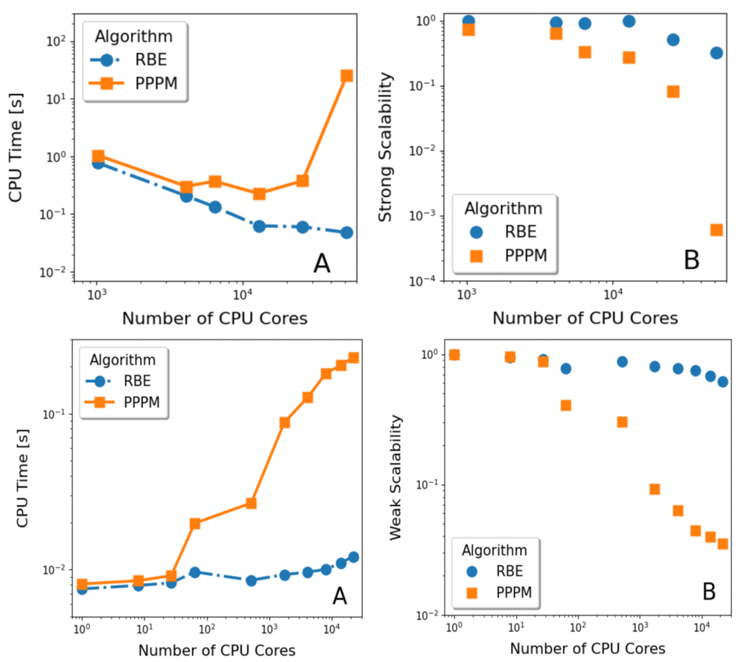
LAMMPS-RBE在“思源一号”上的测试结果
值得一提的是,洪亮教授团队将该算法应用于计算药物分子和靶蛋白的结合自由能(Free energy pertubation,简称FEP),在多个靶点验证了算法的精度,与行业头部企业薛定谔相当。
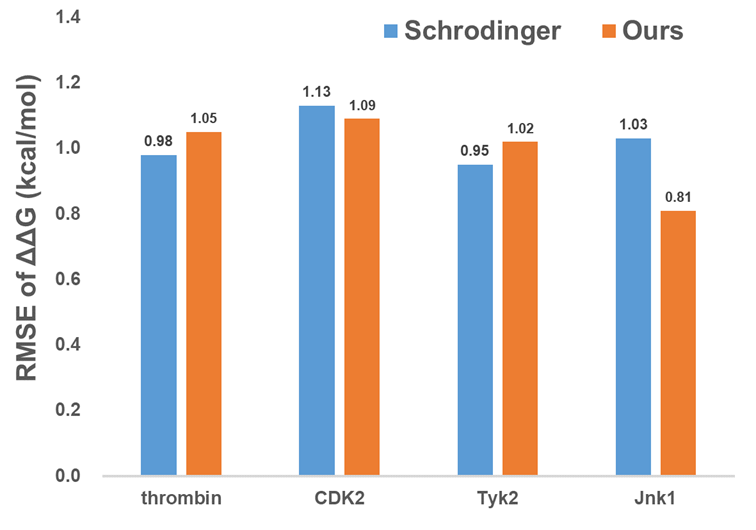
FEP计算中,洪亮教授团队与薛定谔的算法精度比较
团队目前正致力于解决RBM算法在生物体系动力学模拟中的应用,具体涉及短程相互作用的精准加速计算,以克服传统分子动力学模拟在计算相互作用时消耗大量时间的问题。在此基础上,结合GPU大装置、创新计算化学和计算物理方法,有望实现受体-配体结合动力学的加速模拟,尤其是超大生物体系的模拟。
参考文献
- Shi Jin, Lei Li and Jian-Guo Liu, Random Batch Methods (RBM) for interacting particle systems, J. Comp. Phys. 400, 108877, 2020.
- S. Jin, L. Li, Z. Xu and Y. Zhao, A random batch Ewald method for particle systems with Coulomb interactions, SIAM J. Sci. Comput., 43 (2021), B937-B960.
- J. Liang, P. Tan, Y. Zhao, L. Li, S. Jin, L. Hong and Z. Xu, Superscalability of the random batch Ewald method, J. Chem. Phys., 156 (2022), 014114.
- J. Liang, Z. Xu and Y. Zhao, Random-batch list algorithm for short-range molecular dynamics simulations, J. Chem. Phys., 155 (2021), 044108.
- J. Liang, P. Tan, L. Hong, S. Jin, Z. Xu and L. Li, A random batch Ewald method for charged particles in the isothermal-isobaric ensemble, arXiv: 2110.14362.




